6.8 GSEA富集分析 EMP_GSEA_analysis
基因集富集分析(Gene Set EnrichmentAnalysis, GSEA)的基本思想是将待分析基因集的基因与预先定义的基因集的基因(通常来自功能注释或先前实验的结果)进行比较,将待分析基因按照计算值从高到低排序(即:排序表),判断待分析基因在预先定义的基因集里面是随机分布还是主要聚集在顶部或是底部。如果是聚集在顶部或是底部,则说明预先定义的基因集对于表型的差异有贡献。GSEA的排序方法包括三种:基于信噪比、基于相关性结果和基于差异性分析结果。
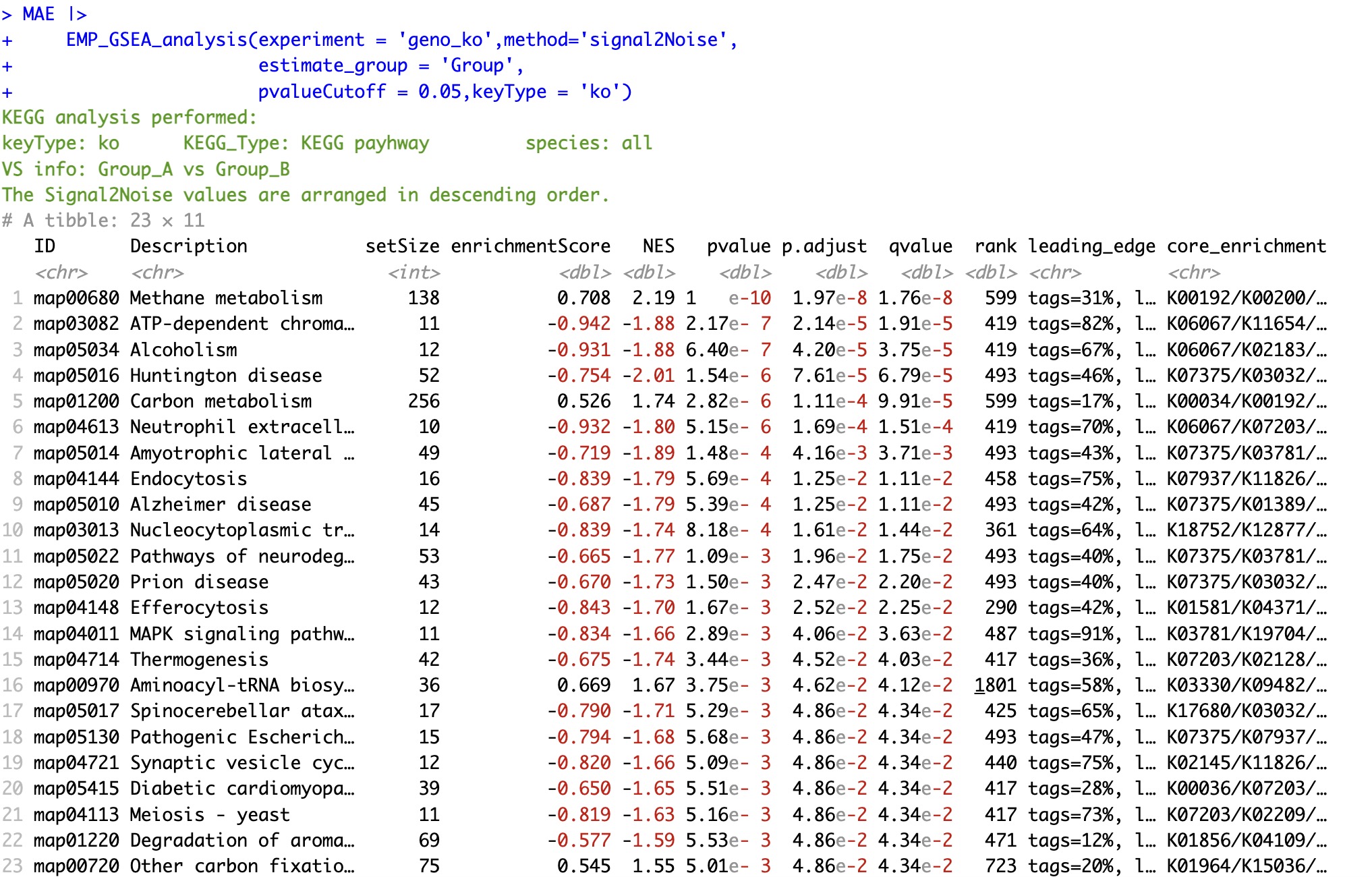
6.8.1 基于信噪比signal2Noise排序
signal2Noise是GSEA官网提供的计算方式,用于对待分析基因进行排序。
🏷️示例:
MAE |>
EMP_GSEA_analysis(experiment = 'geno_ko',method='signal2Noise',
estimate_group = 'Group',
pvalueCutoff = 0.05,keyType = 'ko')
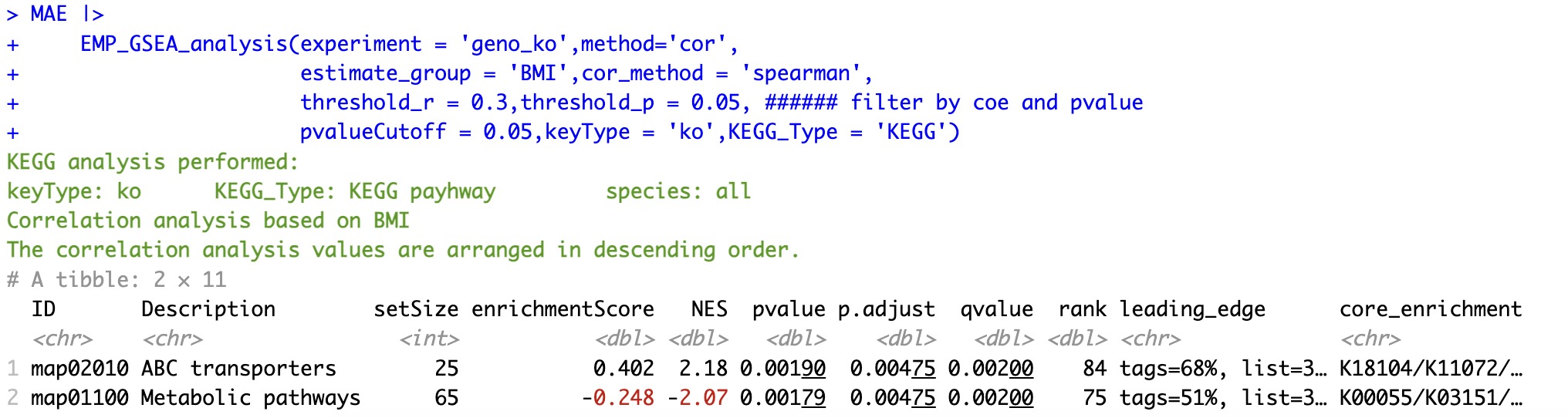
6.8.2 基于相关性排序
在GSEA的相关性排序算法中,可以通过指定参数threshold_r(相关系数的绝对值阈值)和threshold_p(p值阈值)进行过滤。具体而言,如果某个待分析基因与coldata中的特征的相关系数绝对值低于指定阈值,或者其p值大于指定阈值,那么该基因将会被从排序表中剔除。
🏷️示例:
MAE |>
EMP_GSEA_analysis(experiment = 'geno_ko',method='cor',
estimate_group = 'BMI',cor_method = 'spearman',
threshold_r = 0.3,threshold_p = 0.05, ###### filter by coe and pvalue
pvalueCutoff = 0.05,keyType = 'ko',KEGG_Type = 'KEGG')
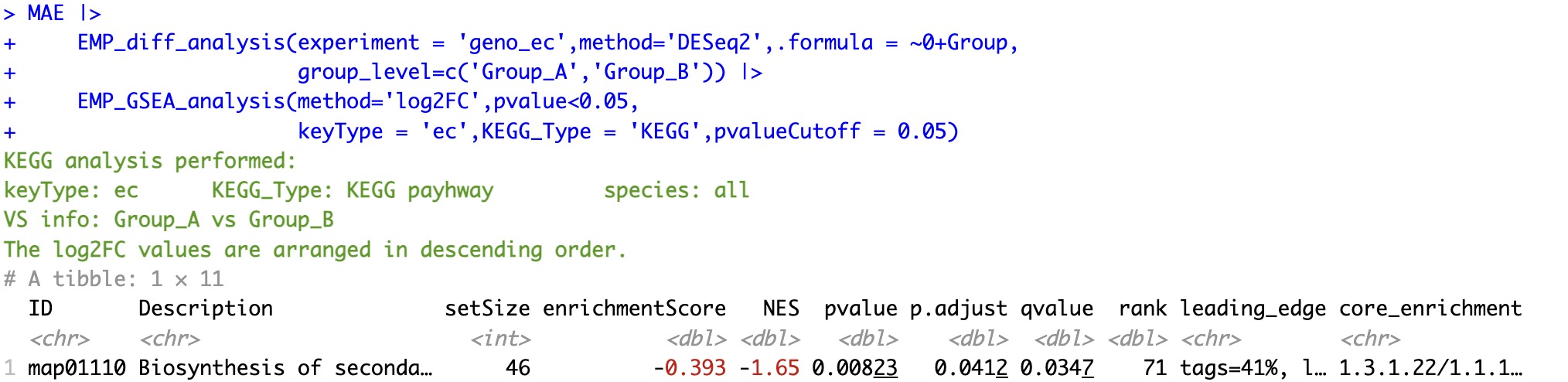
6.8.3 基于差异性分析log2FC排序
注意:
①必须先使用模块EMP_diff_analysis进行差异性分析(分组只能为两组,否则无法输出可用于对接模块EMP_GSEA_analysis的结果),然后再使用模块EMP_GSEA_analysis指定参数method='log2FC'做富集分析。
②需区分模块EMP_GSEA_analysis中的参数pvalue和pvalueCutoff,前者对应模块EMP_diff_analysis分析结果中的pvalue,后者对应模块EMP_GSEA_analysis分析结果中的校正p值(例如:fdr、bonferroni等)。
①必须先使用模块EMP_diff_analysis进行差异性分析(分组只能为两组,否则无法输出可用于对接模块EMP_GSEA_analysis的结果),然后再使用模块EMP_GSEA_analysis指定参数method='log2FC'做富集分析。
②需区分模块EMP_GSEA_analysis中的参数pvalue和pvalueCutoff,前者对应模块EMP_diff_analysis分析结果中的pvalue,后者对应模块EMP_GSEA_analysis分析结果中的校正p值(例如:fdr、bonferroni等)。
🏷️示例:
MAE |>
EMP_diff_analysis(experiment = 'geno_ec',method='DESeq2',.formula = ~0+Group,
group_level=c('Group_A','Group_B')) |>
EMP_GSEA_analysis(method='log2FC',pvalue<0.05,
keyType = 'ec',KEGG_Type = 'KEGG',pvalueCutoff = 0.05)
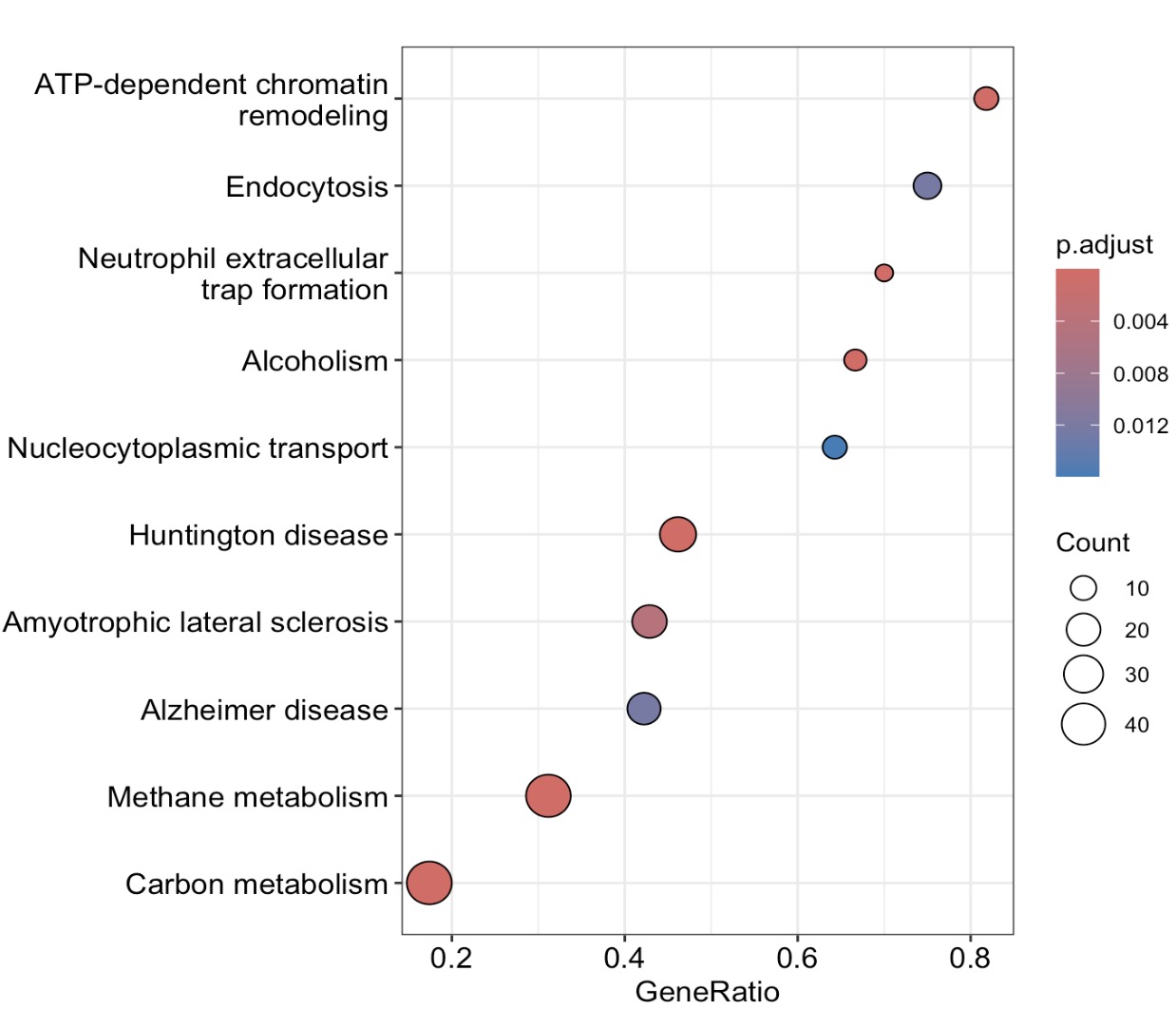
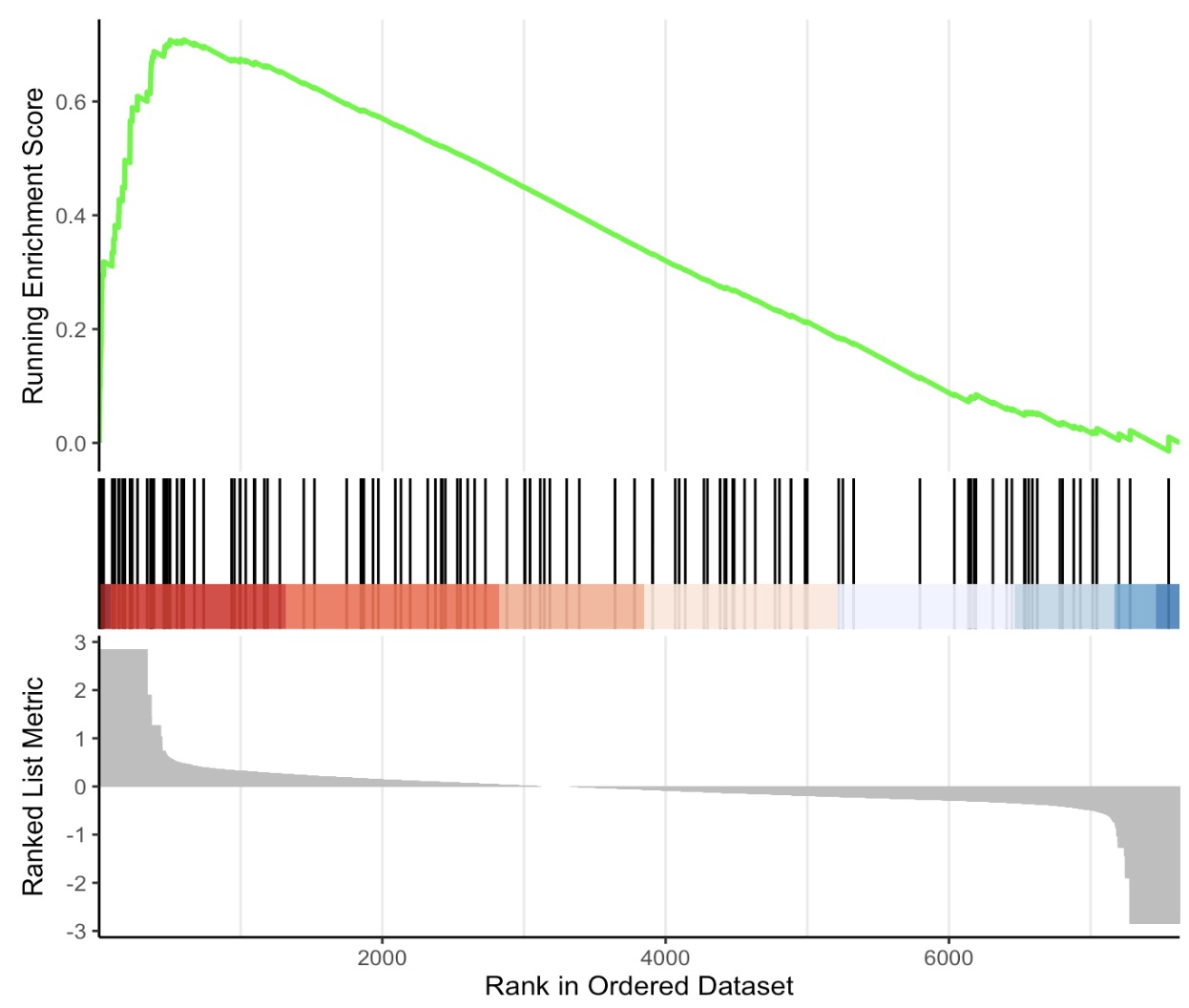
6.8.4 GSEA的可视化
🏷️示例:
MAE |>
EMP_GSEA_analysis(experiment = 'geno_ko',method='signal2Noise',
estimate_group = 'Group',
pvalueCutoff = 0.05,keyType = 'ko') |>
EMP_GSEA_plot(geneSetID='map00680')
MAE |>
EMP_GSEA_analysis(experiment = 'geno_ko',method='signal2Noise',
estimate_group = 'Group',
pvalueCutoff = 0.05,keyType = 'ko') |>
EMP_enrich_dotplot()